25. Anorganische Chemische Verbindungen
Contents
Nachdem wir letztes Mal über störungssichere Übertragung gesprochen haben, wollen wir heute systematische (anorganische) Chemie betreiben.
0. Unser Ziel
Wir haben 2 Teilziele. Zum einen wollen wir die (meisten) Elemente des Periodensystems systematisch erfassen, also die hier:
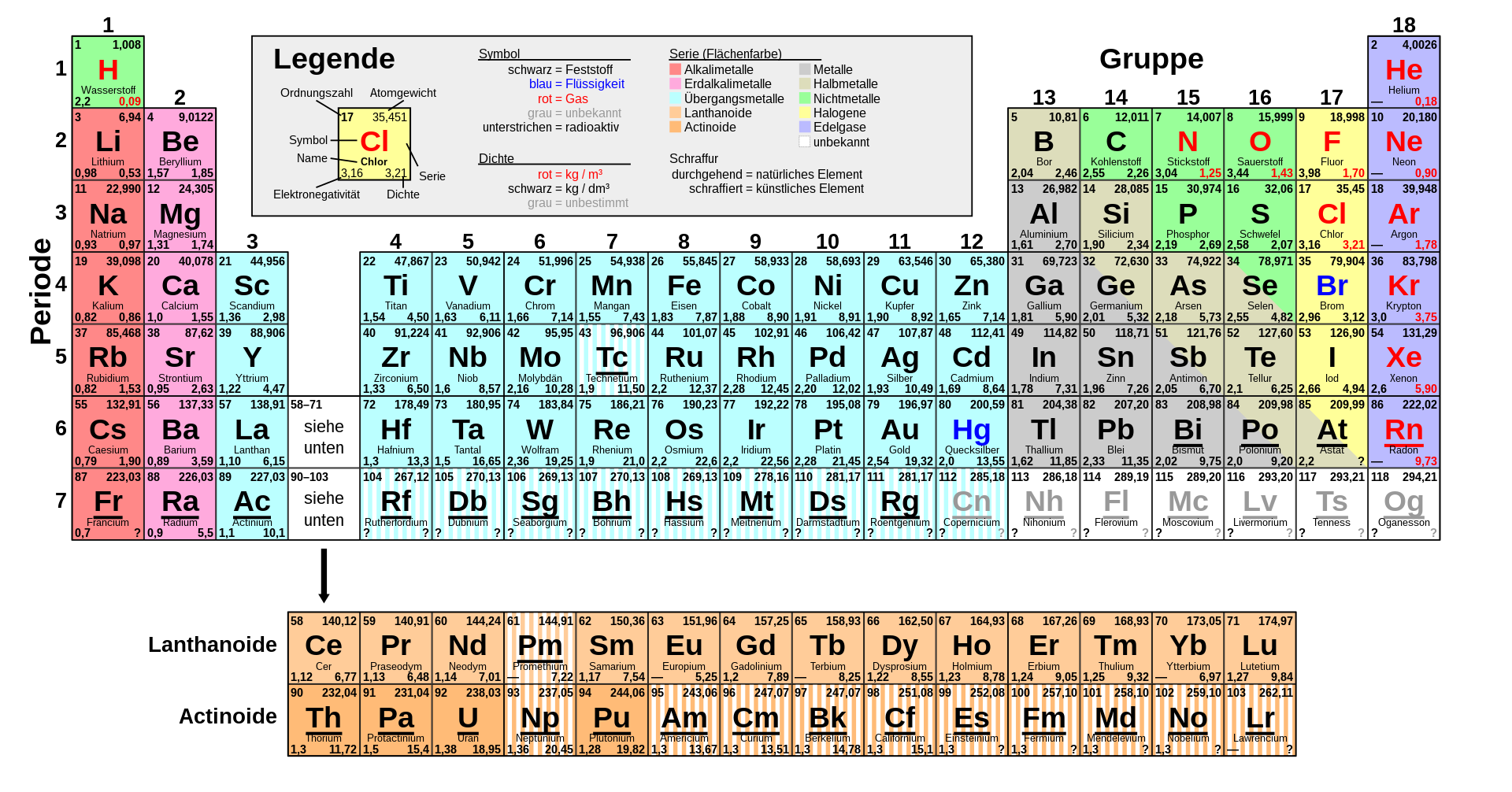 Bild 1: Periodensystem der Elemente. CC Antonsusi 2019.
Bild 1: Periodensystem der Elemente. CC Antonsusi 2019.
Und zum anderen wollen wir die Summenformeln von (einfachen) anorganischen Verbindungen ausrechnen, also so etwas wie NaCl oder Al$_2$O$_3$.
1. chemische Elemente darstellen
Wie man aus den Farben des obigen Periodensystems erkennt, kommen die Elemente in ca. 4 Klassen: Metalle, Halbmetalle, Nichtmetalle, Edelgase. Da es nur ca. 118 Elemente gibt und keine neuen einfach erzeugt werden können, wollen wir die Elemente als Enums darstellen. Entsprechend der 4 Klassen wollen wir 4 verschiedene Enums anlegen. Trotzdem sollen alle gleichwertig als Atome erkennbar sein.
Als ersten Ansatz stellen wir die Atome durch ein Interface dar. Jede konkrete Klasse (auch Enums) kann dieses Interface implementieren und die Bindungsfunktionen können dann ein Atom entgegen nehmen. Vielleicht etwa so:
|
|
Wie kann man nun die konkreten Elemente eintragen? Etwa so:
|
|
Die Edelgase haben einige Gemeinsamkeiten:
- keine Valenzen,
- Symbol meist aus den ersten 2 Buchstaben.
Deshalb habe ich die im Konstruktor bereits eingetragen und die Default-Argumente nach hinten verschoben (sodass man die notwendigen Argumente am Anfang ohne Namen angeben kann).
Ganz ähnlich können wir mit den Nichtmetallen verfahren:
|
|
Hier liegen verschiedene Valenzen vor, sodass wir die nicht mehr standardmäßig initialisieren können. Viele Symbole leiten sich aber aus dem ersten Buchstaben ab, sodass ich das Symbol unten berechnet habe und nur bei Chlor, Brom und Selen überschieben habe (dort besteht es aus 2 Buchstaben).
Ich denke, dass das Prinzip jetzt klar geworden ist. Hier noch die Halbleiter/Halbmetalle:
|
|
Am Aufwändigsten ist die Klasse der Metalle, weil die meisten chemischen Elemente Metalle sind. Ich gebe die hier nur ausschnittsweise an, um auf ein paar Besonderheiten hinzuweisen:
|
|
Die restlichen Daten kann man der Wikipedia entnehmen.
Um die Daten zu testen, schreiben wir ein kleines Hauptprogramm:
|
|
Allerdings brauchen wir noch 2 Methoden für Atome:
|
|
Jetzt sollte die Ausgabe des Testprogramms etwa wie folgt aussehen:
|
|
2. 2-Elementige Verbindungen
Dazu führen wir die Klasse data class Bond2(val element1 :Atom, val element2 :Atom) ein. Aber offenbar brauchen wir noch 2 weitere Informationen: Wieviele Atomrümpfe der jeweiligen Art machen ein neutrales Gebilde aus? Also
|
|
Um eine Verbindung zu erzeugen, können wir entweder die Atomrümpfe und Stöchiometrien selbst angeben oder wir schreiben uns eine Funktion, die die ausrechnet. Letzteres kann man mit den gegebenen Daten wie folgt tun:
|
|
Allerdings müssen wir noch 3 Funktionen implementieren:
|
|
Was bedeuten die 3 Funktionen?
Der Betrag heißt im Englischen absolute value, abgekürzt mit abs. Aus der Definition sieht man, dass der Betrag einer großen ganzen Zahl eine nicht-negative große ganze Zahl ist.
gcd steht für größter gemeinsamer Teiler (engl. greatest common divisor) und den brauchen wir, um lcm zu berechnen.
2.2 Was ist nun lcm und was hat das mit Stöchiometrie zu tun?
Betrachten wir 2 Elemente wie etwa Aluminium (Al) und Sauerstoff (O). Ersteres hat die Valenz +3 und letzteres die Valenz -2. Wie müssen die also zusammenpassen?
- eine Valenz muss negativ sein, die andere positiv (also das Produkt negativ);
- wir müssen das kleinste gemeinsame Vielfache bilden, englisch least common multiple, und das macht die Funktion
lcm; - die Stoffmengen ergeben sich aus diesem
lcmgeteilt durch den Betrag der Valenz.
2.3 Funktioniert das alles?
Um das zu testen, können wir entweder wieder ein Hauptprogramm schreiben oder eine Testbibliothek verwenden. Der Vorteil des letzteren ist, dass wir Zusicherungen (engl. assertions) überprüfen können und die Bibliothek sagt uns dann, wo etwas schiefgegangen ist.
Dazu müssten wir ggf. die Kompiler-Konfiguration anpassen, also die Datei “build.gradle.kts” (im Hauptverzeichnis des Projektes). Der Abschnitt dependencies sollte folgende 3 Zeilen enthalten:
|
|
Sowie auch ein zusätzlicher Abschnitt tasks.test mit diesem Eintrag vorhanden sein. Falls du etwas geändert hast, erscheint oben rechts ein kleines Symbol mit einem grauen Elefanten und einem blauen Kreisel. Dieses Symbol musst du klicken, damit die veränderte Konfiguration importiert wird. Falls das Symbol mal nicht auftauchen sollte, kannst du auch rechts den Reiter “Gradle” (ebenfalls mit einem grauen Elefanten) öffnen und dort oben links im Unterfenster auf den grauen Kreisel (2 Pfeile, die im Kreis zeigen) klicken. Danach kannst du das Gradle-Unterfenster und auch den Editor für die “build.gradle.kts” wieder schließen.
Es sollte links im Projektbaum unter Quellen “src” ein zweites Verzeichnis “test/kotlin” geben. In diesem Verzeichnis legst du jetzt eine neue Kotlin Klasse “Bond2Tester” an: Dazu im Projektbaum (links) den Reiter “test” aufklappen und dort auf “kotlin” mit der rechten Maustaste klicken. Im Menü sollte der Punkt “Neu…” und darunter der Punkt “Kotlin Klasse/Datei” erscheinen, den du auswählst. Danach sollte ein fliegendes Eingabefenster aufgehen, in dem du oben den Namen “Bond2Tester” eingibst. Unten sollte bereits “Klasse” ausgewählt sein, sodass du einfach <Enter> drücken kannst.
Danach sollte sich ein neues Unterfenster im Editor öffnen, in dem eine Klasse class Bond2Tester {} angelegt ist. Über dieser Klasse fügst du einen einzelnen Import ein: import kotlin.test.* (auf einer eigenen Zeile, ggf dahinter noch 1 Zeile freilassen).
Jetzt sind wir bereit, den ersten Test anzulegen:
In der Klasse Bond2Tester schreibst du auf eine neue Zeile @Te, dann sollte das Vorschlagsmenü aufgehen und die Zeile @Test fun anbieten, damit geht es weiter. Der Test sieht also wie folgt aus:
|
|
Um zu sehen, was der Test produziert, kannst du links im Editor auf den Doppelpfeil vor der Klasse rechts-klicken und aus dem Menü “Run ‘Bond2Tester’” auswählen. Danach sollte sich unter dem Editor ein Unterfenster öffnen, in dem du nach kurzer Übersetzungszeit rechts nur “BUILD SUCCESSFUL in …s” und links einen grünen Haken mit “Test Results” siehst. Wenn du links auf das Dreieck klickst, dann öffnet sich ein Unterzweig, in dem du die Klasse “Bond2Tester” sehen solltest (auch mit einem grünen Haken davor). Auch hier auf das graue Dreieck klicken. Dann sollest du einen dritten Haken mit dem Namen “metalOxides” sehen. Wenn du auf diesen Namen klickst, kannst du im rechten Unterfenster die Metalloxide sehen: Die Metalle sind der Reihe nach aufgelistet, jeweils vorn der Name und dahinter eine Liste (eigentlich eine Menge), in der aber nur kryptische Namen “Bond2@2f23ab” stehen. Um hier die Summenformel auszugeben, müssen wir noch die Klasse Bond2 mit einer sinnvollen fun toString() versehen. Dazu fügst du in die Klasse folgende Methode ein:
|
|
Um den Test erneut zu starten, musst du einfach auf den grünen Pfeil oberhalb des Editors klicken. Jetzt sollten schon viele sinnvolle Summenformeln dastehen, z.B.
|
|
Falls dich die 1en auch stören, müssten wir die toString()-Methode wie folgt anpassen:
|
|
D.h. dass die Konvention unter Chemikern ist, wenn die Stoffmenge 1 ist, dann wird die weggelassen.
Außerdem verwendet man in der Summenformel üblicher Weise die Kurzsymbole. Dazu passen wir die toString()-Methode der Enums an:
|
|
Jetzt kannst du erneut testen.
2.4 Nichtmetall-Oxide
Auch die kann unser Programm bereits. Dazu musst du einfach eine 2. Testfunktion schreiben, wie folgt:
|
|
2.5 Keine Verbindungen mit Edelgasen
Auch das sollte unser Programm beherrschen. Dazu schreiben wir folgende Testfunktion:
|
|
Hier habe ich jetzt etwas Neues eingebaut. Wir wollen nicht nur einfach die Ergebnisse anzeigen, sondern wir wollen auch sicherstellen, dass es da keine Verbindungen gibt.
Deshalb haben wir die Zeile assertEquals(expected, actual) hinzugefügt, wobei wir emptySet() erwarten (engl. expected) und bonds als aktuellen Wert (engl. actual) übergeben.
Wenn du das Programm ausführst, sollten inzwischen 3 grüne Haken unter der Klasse Bond2Tester erscheinen.
Wenn du sicherstellen willst, dass das auch wirklich testet, kannst du die Spezifikation für Helium abändern, etwa wie folgt:
|
|
Wenn du jetzt die Tests wieder ausführst, solltest du statt einem grünen Haken ein gelbes Kreuz sehen. (Falls du sogar ein Ausrufezeichen in einem roten Kreis siehst, ist im Programm etwas gewaltig schiefgegangen.) Wenn du im Testfenster links auf den fehlgeschlagenen Test klickst, solltest du rechts eine Beschreibung des Fehlers sehen, leider in Englisch. Bei mir sieht das etwa so aus:
|
|
Die erste Zeile kommt vom erfolgreichen Teil des Tests, d.h. er konnte irgendwelche Verbindungen bestimmen und hat diese ausgegeben (zusammen mit dem Namen Helium).
Dann steht da expected: ... but was: ... Das bedeutet, dass etwas nicht so war, wie es erwartet wurde. Wenn du in der Fehlermeldung weiter unten suchst, dann gibt es eine Zeile, in der etwas blau markiert ist “Bond2Tester.kt:20”. Wenn du auf diesen blauen Text klickst, kommst du direkt zu der Stelle, an der etwas schiefging. Das sollte die Zeile mit dem assertEquals sein. Am Anfang der Fehlermeldung steht auch, was schiefgegangen ist, nämlich, dass keine Verbindungen erwartet wurden, es aber welche gab.
Wenn du nun die Spezifikation von Helium wieder zurück änderst und den Test erneut laufen lässt, dann sollten wieder alle Tests grün sein.
2.6 Halbmetall-Oxide
Auch Halbmetalle lassen sich oxidieren:
|
|
2.7 Salze
Auch die kann unsere Methode bereits. Dazu müssen wir nur noch auswählen, welche Elemente sich mit Metallen zu Salzen verbinden können:
|
|
Das sind ziemlich viele, und die können anhand der Valenzen alle stöchiometrisch korrekt gebildet werden.
2.8 Halbmetall-Verbindungen
Etwas schwieriger sieht es bei den Halbmetall-Verbindungen aus. Das Programm kann nicht erkennen, wenn es Verbindungen, die stöchiometrisch funktionieren, aus Stabilitätsgründen gar nicht gibt. Deshalb schränken wir den Test ein:
|
|
3. Säuren und Basen
Wie kann man jetzt Säuren und Basen darstellen?
Dazu brauchen wir offenbar auch eine Klasse Bond3, die aus 3 Atomen zusammengesetzt ist, etwa wie folgt:
|
|
Und dann müssen wir eine Funktion zum Reagieren mit Wasser schreiben (englisch hydrate). Allerdings gibt es noch 1 Problem: Die Basen bestehen meist aus 3 Elementen, z.B. NaOH, aber Säuren können aus 2 oder aus 3 Elementen bestehen. Deshalb wollen wir den Rückgabetyp List<Bond> schreiben, wobei Bond dann entweder Bond2 oder Bond3 sein kann. Was kann man da machen?
Die Lösung ist oben bereits angedeutet: Wir schreiben ein weiteres Interface:
|
|
Damit das Ganze funktioniert, müssen wir auch die Klasse Bond2 entsprechend anpassen:
|
|
Jetzt können wir die Methode implementieren:
|
|
Ok, also für die Metalle und Halbmetalle ist es nicht so schwer, sie bilden einfach Hydroxide entsprechend ihrer Valenzen.
Für die Halogene unterscheidet es sich in Fluor und die restlichen (außer Astat, welches ich einfach als “Halbmetall” eingeteilt habe). Bei Fluor gibt es nur Fluorwasserstoff, dessen Bindung(en) wir bereits mit bind(...) berechnen können (das Ergebnis ist ein Bond2). Für die anderen gibt es noch die chlorige Säure, Chlorsäure und Perchlorsäure. In diesen hat das Halogen eine positive Valenz. Deshalb wird als erstes auf { it>0 } gefiltert. Entsprechendes gibt es für Brom und Iod.
Für die restlichen Elemente der Sauerstoffgruppe gibt es neben dem Schwefelwasserstoff noch 2 mögliche 3er Bindungen: Schwefelige Säure (H$_2$SO$_3$) und Schwefelsäure (H$_2$SO$_4$), und entsprechendes für Selen.
Für Sticksoff gibt es neben NH$_3$ (Ammoniak) noch die salpetrige Säure (HNO$_2$) und die Salpetersäure (HNO$_3$). Für Phosphor gibt es neben Phosphin (PH$_3$) noch hypophosphorige Säure (H$_3$PO$_2$), phosphorige Säure (H$_3$PO$_3$) und Phosphorsäure (H$_3$PO$_4$).
Schließlich gibt es für Kohlenstoff nur die Kohlensäure. Diese 3 Teile müssen wir noch ergänzen:
|
|
Jetzt kannst du das testen:
|
|
Im Prinzip kannst du auch noch einen Test schreiben, dass sich Edelgase nicht hydratisieren lassen.
3.2 Was ist Säure und was ist Base?
Im Prinzip kann man das auch noch entscheiden. Dazu müssen wir aber das Interface Bond modifizieren, etwa wie folgt:
|
|
Wenn du jetzt die toString()-Methoden abänderst, sodass sie den Bindungstyp in Klammern schreiben, dann kannst du die Unterscheidung im Test sehen:
|
|
9. Selber Probieren
Uff, das war eine ganz schöne Fleißarbeit. Tja, Chemie ist viel Systematik, aber auch einige Ausnahmen.
Ich hoffe, dass du die Programme selbst probiert hast.
9.1 Organische Chemie
Neben der anorganischen Chemie gibt es auch die organische Chemie. Hier kannst du vor allem mit den Alkanen anfangen, also Methan, Ethan, Propan, Butan, … .
Als nächstes kannst du die Derivate bilden, also Säuren, Alkohole, Aldehyde, Alkene, Alkine.
Und dich schließlich mit Liganden (Resten, engl. moiety) versuchen.
Viel Spaß beim Probieren.